Tabela de conteúdos
Introdução à ordenação multivariada
Ordenação é um método de redescrição dos dados multivariados de forma a apresentá-los em poucas dimensões, geralmente 2 ou 3, com a menor perda possível de informação. Veja o exemplo da representação gráfica de duas parcelas em um espaço dimensional de duas espécies.
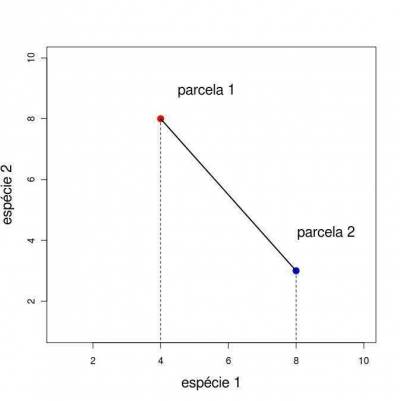
Quando temos apenas duas dimensões de atributos (no caso duas espécies) a representação gráfica dos objetos, no nosso caso as parcelas, é direta.
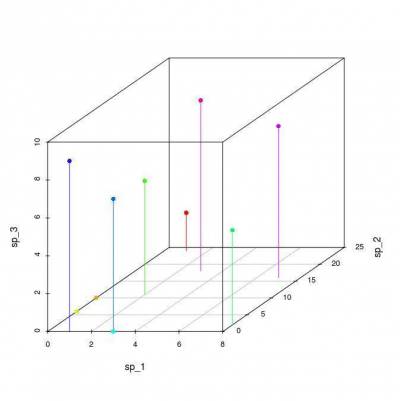
Conforme vamos acrescentando informações de novos atributos (espécies) aos nossos objetos (parcelas), a representação gráfica torna-se mais difícil. Com três dimensões ainda conseguimos representar nossas parcelas em um gráfico.
Os muitos métodos de ordenação como Análise de Componentes Principais (PCA), Análise de Coordenadas Principais (PCO), Análise de Correspondência (CA), têm como objetivo a redução das dimensões descritivas para visualizar melhor as relações entre os objetos, quando são descritos por muitas medidas.
Para seguir em frente é necessário que você tenha na área de trabalho do R as amostras das comunidades virtuais montadas, como descrito no roteiro padrões de gradientes em comunidades.
Certifique-se disso com o comando ls(), que lista os objetos da área de trabalho do R. Na lista resultante devem estar os objetos amost.cont e amost.disc:
> ls() [1] "amost" "amost.cont" "amost.disc"
Ordenação Polar
 Foi um dos primeiros métodos de ordenação e devido a sua simplicidade foi amplamente usado por ecólogos. Apesar de suas limitações e de ter sido preterido por outros métodos mais sofisticados, a Ordenação Polar (OP) pode produzir resultados com interpretação ecológica relevante. Além disso, entender seu algorítmo simples é uma ótima maneira de entender a lógica geral da ordenação.
O objetivo da OP é representar as parcelas em um sistema de coordenadas de forma que a distância entre as parcelas represente suas similaridades e que revelem o gradiente ambiental subjacente. Aqui vamos usar o algoritmo de OP desenvolvido por Bray e Curtis (1957), especificamente para a análise de dados de comunidades de plantas.
Para iniciar a análise, primeiro calculamos a dissimilaridade de Bray-Curtis como nossa medida de distância1).
Foi um dos primeiros métodos de ordenação e devido a sua simplicidade foi amplamente usado por ecólogos. Apesar de suas limitações e de ter sido preterido por outros métodos mais sofisticados, a Ordenação Polar (OP) pode produzir resultados com interpretação ecológica relevante. Além disso, entender seu algorítmo simples é uma ótima maneira de entender a lógica geral da ordenação.
O objetivo da OP é representar as parcelas em um sistema de coordenadas de forma que a distância entre as parcelas represente suas similaridades e que revelem o gradiente ambiental subjacente. Aqui vamos usar o algoritmo de OP desenvolvido por Bray e Curtis (1957), especificamente para a análise de dados de comunidades de plantas.
Para iniciar a análise, primeiro calculamos a dissimilaridade de Bray-Curtis como nossa medida de distância1).
Abaixo uma função da função que calcula a dissimilaridade (distância) de Bary-Curtis entre todos os pares de parcelas. Para usar esta função, copie o código abaixo e cole-a na linha de comando do R.
dis.bc<-function(dados){
nplot=dim(dados)[2]
similar=matrix(NA,ncol=nplot,nrow=nplot)
rownames(similar)<-paste("plot", c(1:nplot))
colnames(similar)<-paste("plot", c(1:nplot))
for(i in 1:(nplot-1)){
m=i+1
for(m in m:nplot){
bc.dist=sum(
abs(dados[,i]-dados[,m]))/(sum (dados[,c(i,m)])
)
similar[m,i]=similar[i,m]=bc.dist
diag(similar)<-0
}
}
return(round(similar,3))
}
Algoritmo da Ordenação Polar
- 1. Use a função acima para produzir a matriz de dissimilaridade para a nossa amostra de comunidades contínuas:
dis1.cont=dis.bc(amost.cont)
- 2. Calcule, para cada uma das parcelas, a soma das distâncias dela em relação às outras parcelas:
somadist1.cont=apply(dis1.cont, 1, sum, na.rm=TRUE) somadist1.cont
- 3. Assinalar a parcela com a maior soma de distâncias 2) e guardar o seu valor para indicar o início do primeiro eixo (x). Essa será nossa primeira referência no espaço de descrição das nossa parcelas (ax).
max(somadist1.cont) nomes.parc = names(somadist1.cont) parc.ax = nomes.parc[somadist1.cont==max(somadist1.cont)][1] parc.ax
- 4. Encontrar agora a parcela que tem a maior distância em relação à primeira referência. Essa será nossa segunda referência no nosso espaço de descrição (bx)
dist.ax=dis1.cont[,parc.ax] dist.ax max.ax=max(dist.ax) max.ax parc.bx=nomes.parc[dist.ax==max.ax] parc.bx
- 4a. Algumas vezes várias parcelas têm valores iguais de distância em relação à parcela que definimos como ax. Nesse caso, ficaremos com aquela que tiver maior soma de distâncias em relação às outras parcelas. Para verificar se este critério de desempate é necessário fazemos assim:
somamax.bx=max(somadist1.cont[parc.bx]) parc.bx=parc.bx[somadist1.cont[parc.bx]==somamax.bx][1] parc.bx
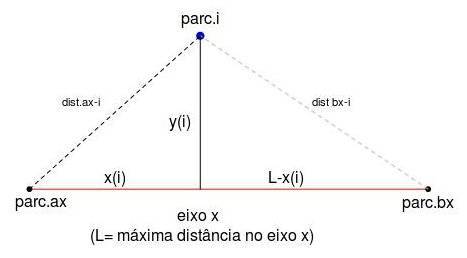
- 5. Agora que temos duas referências do primeiro eixo (parc.ax e parc.bx), podemos calcular a posição de todas as outras parcelas nesse eixo x. Isso funciona como a triangulação que é usada em telemetria para encontrar um animal que está usando um emissor de rádio. Colocando receptores em dois pontos conhecidos e com a informação da distância do emissor (o animal com um rádio colar por exemplo) aos dois receptores, é possível calcular as coordenadas em que o animal está. Veja a representação gráfica do que estamos dizendo:
Para o cálculo utilizamos a equação de Beal (1965) que nada mais é do que resolver a equação da hipotenusa dos dois triângulos acima. Após diversos passos, chegamos finalmente à equação de Beal:
$$ x_i \ = \ \frac{ L^2 \ + \ dist_{ax_i}^2 \ - \ dist_{bx_i}^2 }{2L} $$
onde “L” é a distância entre parc.ax e parc.bx (distância máxima no eixo x), “distax_i” é a distância da parcela “i” em relação à parc.ax, e “distbx_i” é a distância da parcela “i” em relação à parc.bx, conforme pode ser visualizado na figura acima.
- 5a. Agora que entendemos a equação de Beal, antes de aplicá-la, precisamos recuperar os valores de distância das outras parcelas em relação às nossas referências para o primeiro eixo: parc.ax e parc.bx. Para a parc.ax já organizamos em um passo anterior (passo 4) e está no objeto dist.ax. Você pode visualizar esses dados executando a primeira linha do código abaixo. Depois, você pode calcular e inspecionar os valores de distância das outras parcelas em relação à parc.bx, com os dois comandos seguintes:
dist.ax dist.bx=dis1.cont[,parc.bx] dist.bx
- 5b. Agora aplique a equação de Beal a todas as parcelas para encontrar a posição delas no eixo x:
xi = (max.ax^2 + dist.ax^2 - dist.bx^2)/(2*max.ax) xi
- 6. Agora que temos os valores do eixo x de todas as parcelas podemos calcular o valor do segundo eixo dado pelo teorema de pitágoras:
$$ y_i = \sqrt{dist_{ax_i}^2 - x_i^2} $$
yi=sqrt((dist.ax)^2-xi^2) yi
- 6a. Agora fazemos um ajuste do valor da parc.bx no eixo yi, conforme o código abaixo:
yi[parc.bx]=max.ax yi
- 7. Temos agora as coordenadas de cada parcal em dois eixos ortogonais. Vamos organizá-las em um único objeto e fazer um gráfico:
op1.cont=data.frame(xi,yi) op1.cont plot(op1.cont, pch=19, col=rainbow(length(xi)), xlab="Eixo 1", ylab="Eixo 2") text(op1.cont+0.01, labels=rownames(op1.cont))
Função Ordenação Polar
Como somos muito legais, montamos uma função que faz todas essas operações para você não ter que fazer tudo novamente passo a passo. Veja abaixo:
ordena.polar=function(dist)
{
somadist1.cont=apply(dist, 1, sum, na.rm=TRUE) + apply(dist,2,sum, na.rm=TRUE)
nomes.parc=names(somadist1.cont)
parc.ax=nomes.parc[somadist1.cont==max(somadist1.cont)][1]
dist.ax=dist[,parc.ax]
max.ax=max(dist.ax)
parc.bx=nomes.parc[dist.ax==max.ax]
if(length(parc.bx)>1)
{
somamax.bx=max(somadist1.cont[parc.bx])
parc.bx=nomes.parc[somadist1.cont==somamax.bx][1]
parc.bx
}
dist.bx=dist[,parc.bx]
xi= (max.ax^2 + dist.ax^2 - dist.bx^2)/(2*max.ax)
yi=sqrt((dist.ax)^2-xi^2)
yi[parc.bx]=max(dist.ax)
op.xy=data.frame(xi,yi)
opx=jitter(op.xy[,1],10)
opy=jitter(op.xy[,2],10)
plot(opx, opy, pch=19, col=rainbow(length(xi)), xlim=c(-0.1, 1.1), ylim=c(-0.1,1.1), main="Ordenação Polar", sub="Distância Bray-Curtis")
text(opx-0.02,opy-0.02 , labels=paste("p",1:dim(dist)[1], sep=""), cex=0.7)
return(op.xy)
}
Vamos aplicar a função para os dados da comunidade virtual contínua e ver se nossos cálculos estão corretos.
ordena.polar(dis1.cont)
Agora vamos aplicá-la para as comunidades virtuais discretas também.
Primeiro, você precisa usar a função disc.bc para produzir a matriz de dissimilaridade para a nossa amostra de comunidades discretas:
dis1.disc=dis.bc(amost.disc)
Depois, basta aplicar a função ordena.polar na matriz de similaridade das comunidades discretas
ordena.polar(dis1.disc)
Agora é por sua conta!
 Interprete os padrões observados tendo em vista as características que foram utilizadas para construir ambas comunidades.
Interprete os padrões observados tendo em vista as características que foram utilizadas para construir ambas comunidades.
Interpretação de Ordenações
Algumas dicas de interpretação de resultados de ordenações, não só da Ordenação Polar, baseadas no material produzido por Michael Palmer em seu, visualmente não muito atraente, mas ótimo site sobre ordenação:
- A direção dos eixos é arbitrária e não deve afetar a interpretação;
- A escala numérica do eixos não é muito útil para a interpretação (existem algumas exceções como DCA, em que a escala é em unidades de beta diversidade);
- Na OP, assim como muitas outras técnicas de ordenação, a ordem dos eixos é importante. Assim, o eixo 1 é mais importante para a interpretação dos gradientes ambientais que o segundo;
- A experiência prévia do sistema estudado e conhecimento da literatura pertinente são as ferramentas mais poderosas para a interpretação dos gradientes subjacentes ao padrão revelado pela ordenação;
- A interpretação de outros eixos além dos dois primeiros (quando a análise produz) é possível e a decisão de onde parar é uma questão arbitrária e dependente da quantidade e qualidade dos dados. Em algumas técnicas há estatísticas que ajudam a tomar essa decisão;
- É desejável que os eixos não sejam correlacionados, o que é garantido por algumas técnicas. Dessa forma é possível interpretar os eixos como gradientes diferentes.
Depois de passar pelas etapas de construir comunidades virtuais, amostrá-las e aplicar métodos de classificação e ordenação, acreditamos que vocês compreendam os princípios básicos dos métodos analíticos usados para a descrição de comunidades em ecologia. Como dito no início desses roteiros, existem muitos métodos diferentes para conhecer e muitas coisas ainda para resolver em relação a esses métodos. Um vasto mundo interessantíssimo!
Para saber mais
- Ordination Methods for Ecologists, Mike Palmer. (um excelente panorama das técnicas de ordenação e seu uso em ecologia).
- * Clustering and Classification methods for Biologists: site do excelente curso de Alan Fielding.
- Manly, B. 2008. Métodos Estatísticos Multivariados: Uma Introdução. 3 Ed. Artmed, Porto Alegre. (Uma das melhores introduções a técnicas multivariadas para biólogos).
- Prado, P.I. et al. 2002. Ordenação multivariada na ecologia e seu uso em ciências ambientais. Ambiente & Sociedade, (10), 69-83.
- Valentin, J. 2012. Ecologia Numérica: Uma Introdução à Análise Multivariada de Dados Ecológicos. Interciência, Rio de Janeiro. (Outro ótimo texto introdutório)
- Legendre, P., & Legendre, L. 2012. Numerical ecology. Elsevier, Amsterdan. (o livro de refrência de ecologia numérica. Completo e didático, mas é uma leitura mais avançada).